

Biologie moléculaire. Un article de Wikipédia, l'encyclopédie libre.
Pour les articles homonymes, voir BM. La biologie moléculaire est apparue au XXe siècle, à la suite de l'élaboration des lois de la génétique, la découverte des chromosomes et l'identification de l'ADN comme support chimique de l'information génétique. Histoire[modifier | modifier le code] La biologie moléculaire est apparue dans les années 1930, le terme n'ayant cependant été inventé qu'en 1938 par Warren Weaver. Warren Weaver était à l'époque directeur des Sciences Naturelles pour la Fondation Rockefeller et pensait que la biologie était sur le point de vivre une période de changements significatifs étant données les avancées récentes dans les domaines tels que la diffractométrie de rayons X.
Relation avec les autres sciences biologiques « à l'échelle moléculaire »[modifier | modifier le code] Relation schématique entre la biochimie (biochemistry), la génétique (genetics) et la biologie moléculaire (molecular biology) Réaction en chaîne par polymérase. Un article de Wikipédia, l'encyclopédie libre.
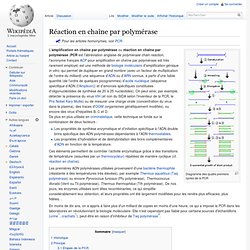
Pour les articles homonymes, voir PCR. Diagramme des quatre premiers cycles de la PCR L'amplification en chaîne par polymérase ou réaction en chaîne par polymérase (PCR est l'abréviation anglaise de polymerase chain reaction, l'acronyme français ACP pour amplification en chaîne par polymérase est très rarement employé) est une méthode de biologie moléculaire d'amplification génique in vitro, qui permet de dupliquer en grand nombre (avec un facteur de multiplication de l'ordre du milliard) une séquence d'ADN ou d'ARN connue, à partir d'une faible quantité (de l'ordre de quelques picogrammes) d'acide nucléique (séquence spécifique d’ADN (l’Amplicon)) et d'amorces spécifiques constituées d'oligonucléotides de synthèse de 20 à 25 nucléotides).
Ces éléments permettent de contrôler l’activité enzymatique grâce à des transitions de température (assurées par un thermocycleur) répétées de manière cyclique (cf. réaction en chaîne). Idem au cycle 2. Électrophorèse. Un article de Wikipédia, l'encyclopédie libre.
Technicienne utilisant l'électrophorèse pour séparer des protéines. L’électrophorèse est — avec la chromatographie — la principale des techniques utilisées en biologie pour la séparation et la caractérisation des molécules. Elle a quelques applications en chimie, mais est principalement utilisée en biochimie ou biologie moléculaire pour la séparation des protéines ou des acides nucléiques. Dans un milieu donné, la séparation des particules se fait en fonction de leur charge électrique et pour des charges identiques, en fonction de leur taille. Southern blot. Un article de Wikipédia, l'encyclopédie libre.
Un transfert d'ADN[1] est une méthode de biologie moléculaire permettant l'analyse de l'ADN. Elle a été inventée par Edwin Southern, un professeur britannique de biologie moléculaire. C'est ce nom (Southern blot) qui a, par jeu de mot, inspiré l'appellation d'autres techniques : western blot, northern blot et Far-eastern blot. Principe[modifier | modifier le code] Nous partons des fragments d'ADN obtenus par les enzymes de restriction. Méthode[modifier | modifier le code] L'ADN est digéré par les enzymes de restriction.Les fragments d'ADN sont placés dans les puits d'un gel d'électrophorèse (gel d'agarose à faible concentration --> 0,6 à 0,8 %).Le gel est alors mis dans une cuve horizontale contenant une solution tampon et deux électrodes (+ et -).
Résultat[modifier | modifier le code] La sonde va révéler la position sur la membrane des fragments d'ADN recherchés. Notes et références[modifier | modifier le code] Animation expliquant la technique. Northern blot. Un article de Wikipédia, l'encyclopédie libre.
Un transfert d'ARN[1] (également appelé northern blot (en) ou buvardage de northern), est une méthode de biologie moléculaire permettant l'analyse de l'ARN. Elle dérive du Southern blot (appelé ainsi par le biologiste Edwin Southern qui a aussi inspiré le nom western blot qui fait référence aux séquences en acides aminés des protéines), sauf qu'au lieu d'étudier de l'ADN, on étudie de l'ARN. L'ARN va être analysé par électrophorèse, permettant de séparer les ARN en fonction de leur taille. Puis ils sont détectés par une sonde. Western blot. Un article de Wikipédia, l'encyclopédie libre.
Un transfert de protéines[1] (également appelé western blot ou buvardage de western[2]), est une méthode de biologie moléculaire permettant la détection et l'identification de protéines spécifiques dans un échantillon biologique (sérum ou autre extrait ou homogénat tissulaire). C'est un outil de diagnostic complémentaire. Technique[modifier | modifier le code] Cette technique née des progrès de la protéomique, de la biologie moléculaire et de l'Immunofluorescence utilise l'électrophorèse sur gel de polyacrylamide pour séparer des protéines, préalablement dénaturées, selon leur masse.
Ces protéines sont ensuite transférées depuis le gel sur une membrane (typiquement en nitrocellulose), où elles sont exposées à un anticorps spécifique de la protéine d'intérêt. La méthode fut mise au point dans le laboratoire de George Stark à Stanford. Puce à ADN. Un article de Wikipédia, l'encyclopédie libre.
Principe d'utilisation de la puce à ADN. Les puces à ADN sont aussi appelées puces à gènes, biopuces, ou par les termes anglais « DNA chip, DNA-microarray, biochip ». Les termes français microréseau d'ADN et micromatrice d'ADN sont aussi des termes proposés par l'Office québécois de la langue française[1]. Principe[modifier | modifier le code] Par exemple on peut marquer l'ADN complémentaire du malade en vert et du traité en rouge, ou bien, du témoin en rouge et du traité en vert. Les molécules d'ADN fixées sur la lame sont appelées des sondes même si la nomenclature peut varier. Utilisation[modifier | modifier le code] Généralement, après une étude de puce à ADN, la bio-informatique extrait une liste de gènes intéressants (en fonction de ce que l'on cherche).